在生物信息学的世界里,单细胞数据分析一直是科研人员关注的热点领域。小明作为一名热爱探索的科研工作者,在研究过程中接触到了一种强大的工具——SCENIC/pySCENIC。今天,他将通过自己的视角,带领大家深入了解如何利用这个工具完成单细胞数据调控网络分析。
什么是SCENIC/pySCENIC?
SCENIC(Single-Cell Regulatory Network Inference and Clustering)是一种用于推断单细胞水平基因调控网络的强大工具。它能够帮助我们识别潜在的转录因子及其靶基因,并构建出精确的调控网络模型。pySCENIC则是其Python版本,功能强大且易于操作。
第一步:安装与准备
小明首先需要确保环境配置无误。他使用Anaconda创建了一个全新的虚拟环境,并按照官方文档中的步骤依次安装了pySCENIC及相关依赖库。
具体命令如下:
conda create -n pyscenic_env python=3.8
conda activate pyscenic_env
pip install loompy scvelo pyscenic安装完成后,他还下载了所需的参考数据库,如cisTarget数据库,这是后续分析的重要基础。
第二步:数据预处理
接下来,小明开始对原始单细胞RNA测序数据进行预处理。他选择了一种常见的文件格式——Loom文件,这种格式非常适合存储大规模矩阵数据。通过简单的脚本代码,他成功将原始数据转换为适合pySCENIC分析的格式。
以下是关键代码片段:
import loompy
loom_file = 'input_data.loom'
with loompy.connect(loom_file) as ds:
print(ds.shape)经过这一步,数据已经准备好进入下一步分析。
第三步:调控网络推断
在这一环节,小明利用pySCENIC的核心功能,开始构建调控网络。他选择了基于AUCell算法的方法,这是一种评估细胞中特定基因集活性的有效工具。通过运行以下代码,他成功生成了每个细胞中转录因子的活性评分:
from pyscenic.cli.utils import load_signatures
from pyscenic.aucell import aucell
grn = load_signatures('regulons.txt')
auc_mtx = aucell(loom_file, grn)这一过程不仅快速而且准确,为后续分析奠定了坚实的基础。
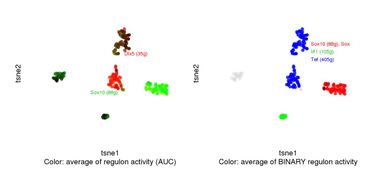
第四步:结果可视化与解读
最后,小明将注意力转向结果的可视化和解读。他使用了多种绘图工具,包括Seaborn和Matplotlib,制作出了精美的热图和散点图。这些图表清晰地展示了不同细胞类型之间的调控差异,为研究提供了重要的线索。
例如,通过热图可以直观看到某些转录因子在特定细胞亚群中的高表达,而散点图则揭示了它们与其他基因之间的关系。
总结来说,小明通过四个简单却高效的步骤完成了单细胞数据调控网络的全流程分析。从环境搭建到最终结果解读,每一步都充满了挑战,但同时也带来了满满的成就感。如果你也对单细胞数据分析感兴趣,不妨尝试一下SCENIC/pySCENIC吧!

发表评论 取消回复